Protein Research Department
Proteine haben vielseitige Funktionen, welche notwendig für das zelluläre Überleben sind. Hierzu gehören z. B. verschiedene Stoffwechsel-, Transport-, und Signaltransduktionswege. Fehlerhafte Proteine, die ihre Aufgabe nicht mehr erfüllen können, verursachen schwerwiegende Erkrankungen. Um die Struktur, Funktionsweise und Netzwerke von Proteinen zu untersuchen, haben sich über 30 Wissenschaftler: Innen der Fakultäten Biologie und Biotechnologie, Chemie und Biochemie, Physik und Astronomie sowie Medizin im Rahmen des Protein Research Departments (PRD) zusammengeschlossen. Die Forschung im PRD befasst sich z. B. mit Fragen, wie einzelne Proteine das Überleben der Zelle beeinflussen, wie Sie sich die Struktur und Funktion von Proteinen entschlüsseln lässt oder wie sich Proteininteraktionen nutzen lassen, um neue Medikamente gegen Erkrankungen zu entwickeln.
Das PRD ist ein interdisziplinäres Forschungsnetzwerk, das hochaktuelle Forschung für ein besseres Verständnis zellulärer Proteinnetzwerke bündelt. Im Mittelpunkt der Forschung des PRD stehen drei Teilbereiche: (A) Biologische Grundlagen der Proteinforschung, (B) Plattformtechnologien in der Proteinforschung, und (C) Translation in die Anwendung. In diesem Netzwerk werden Themen wie Proteinstruktur und -mechanismus, makromolekulare Anordnungen, Funktionen von Membranproteinkomplexen und zelluläres Verhalten aus molekularer Perspektive mit modernsten Methoden aus Strukturbiologie, Biophysik, Biochemie und Zellbiologie untersucht.
Das primäre Ziel des PRD ist es, die Lücke zwischen molekularen und systemischen Ansätzen in der Proteinforschung zu schließen, um zelluläre Prozesse auf molekularer Ebene zu verstehen. Ein quantitatives Verständnis dieser komplexen biologischen Prozesse auf Zellebene setzt fundierte Kenntnisse der unterschiedlichsten genetisch programmierten und dynamisch regulierten Protein-Netzwerken voraus. Im Fokus der Forschung des PRD stehen Prozesse, die Biomembranen involvieren oder von diesen ausgehen. Da Störungen in den beschriebenen Protein-Netzwerken für viele ernsthafte Erkrankungen verantwortlich sind, sollen die gewonnen Erkenntnisse zukünftig in der Entwicklung biotechnologischer Anwendungen mit Langzeitnutzen für die öffentliche Gesundheit resultieren, z.B. in maßgeschneiderten Medikamenten in der Molekulartherapie oder in der Entwicklung von Biomarkern.
Das PRD ist eng mit dem Zentrum für Proteindiagnostik (PRODI; Leiter: Klaus Gerwert) verbunden, das grundlegende Proteinforschung, anwendungsorientierte Diagnostik und Therapieprädikation von neurologischen und onkologischen Erkrankungen vereint. Eine weitere Säule ist das Medical Proteome Center (MPC; Leiterin: Katrin Marcus), welches auf dem Gebiet eines der weltweit führenden Institute ist und state-of-the-art Methoden der Proteomik, Protein-Biochips und innovative Bioinformatik anbietet. Die dritte Säule des PRD ist das Center for System-based Antibiotic Research (CESAR; Leiterin: Julia Bandow), welches sich damit befasst, neue Strukturklassen von Antibiotika zu entwickeln sowie bakterielle Wirkmechanismen und Wirkorte zu identifizieren. Unterstützt wird das PRD zudem durch das DFG-Graduiertenkolleg „Mikrobielle Substratumwandlung“ (MiCon; Sprecher: Franz Narberhaus), das sich mit mechanistischen Grundlagen mikrobieller Stoffwechselvorgänge auf molekularer Ebene beschäftigt, und durch die DFG-Forschergruppe „Struktur und Funktion des peroxisomalen Translokons“ (PerTrans; Sprecher: Ralf Erdmann), die den Mechanismus des Imports gefalteter Proteine durch die peroxisomale Membran aufklären möchte.
Die Promovierenden des PRD sind Mitglieder der International Graduate School of Biosciences (IGB) oder der Graduate School of Chemistry and Biochemistry (GSCB), die beide ein international anerkanntes strukturiertes Doktorandenprogramm in den Proteinwissenschaften anbieten. Die Promovierenden des PRD sind zudem Mitglieder der fakultätsübergreifenden RUB Research School, die ein umfangreiches Soft-Skill-Training zur Optimierung aller Formen von Kommunikation und professionellem Verhalten anbietet.
Protein Forschung in Bochum
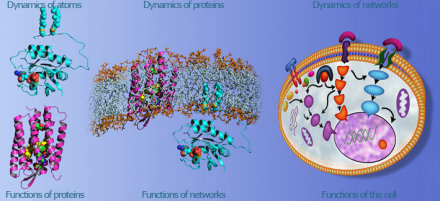
Darstellung der unterschiedlichen Ebenen, auf denen biologische Prozesse im PRD untersucht werden. Ziel ist es, die Lücke zwischen dem detaillierten Verständnis der Struktur und Funktion einzelner Proteine in vitro und dem eher qualitativen Verständnis von Proteininteraktionen in Proteinnetzwerken in vivo sowohl in „bottom-up“ als auch in „top-down“ Ansätzen zu schließen. Dieses Vorgehen erlaubt zum Einen die Identifizierung von Biomarkern zur frühzeitigen Erkennung von Krankheiten, als auch das gezielte Eingreifen in die krankheitsauslösenden Prozesse im Rahmen einer molekularen Therapie.